Forschung
Gehirnzellen vermessen – ohne Skalpell
Wenn die meisten Menschen an MRT denken, sehen sie Bilder des gesamten Gehirns vor sich – Regionen, Bahnen, vielleicht Tumore.
Aber was wäre, wenn man die MRT nutzen könnte, um einzelne Gehirnzellen – also Neuronen und Gliazellen – im lebenden Menschen zu betrachten?
Genau das wollten wir mit unserer aktuellen Studie erreichen: „Resolving Cellular Morphology in the Human Brain with Multiparametric Diffusion MR Spectroscopy“.
Warum wir das tun
Viele neurologische Erkrankungen – Alzheimer, Parkinson, Multiple Sklerose – gehen mit Veränderungen in Form und Größe von Zellen einher, lange bevor auf herkömmlichen MRT-Bildern Gewebeverluste sichtbar sind.
Wir wollten einen Weg finden, diese mikroskopischen Veränderungen nichtinvasiv zu messen.
Den Metaboliten folgen
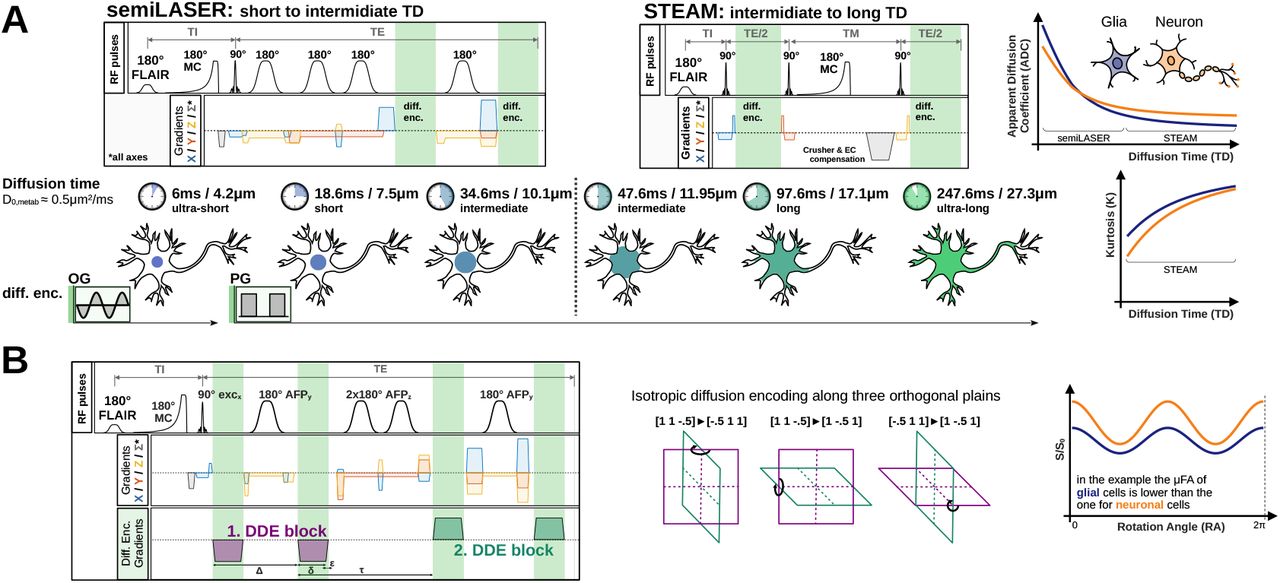
Die klassische Diffusions-MRT misst, wie sich Wassermoleküle im Gewebe bewegen. Doch Wasser befindet sich überall – innerhalb und außerhalb von Zellen – und verrät uns daher nicht, welche Zellen wir eigentlich beobachten.
Unsere Methode, die Diffusions-MR-Spektroskopie (dMRS), verfolgt stattdessen Metaboliten – kleine Moleküle, die sich überwiegend in bestimmten Zelltypen befinden:
- N-Acetylaspartat (NAA): in Neuronen
- Myo-Inositol (mI) und Cholin: in Gliazellen
- Kreatin: in beiden
Indem wir messen, wie sich diese Metaboliten bewegen, können wir Rückschlüsse auf die Größe, Form und Dichte der Zellen ziehen – getrennt für Neuronen und Gliazellen.
Zwei Diffusionsmethoden kombiniert
Wir haben zwei komplementäre MRT-Messungen kombiniert:
- Diffusionszeit-Experimente – sie zeigen, wie weit sich Moleküle in verschiedenen Zeiträumen (6–250 ms) bewegen.
- Doppelte Diffusionskodierung (DDE) – dabei wird der Winkel zwischen zwei Diffusionsgradienten verändert, um zu erkennen, ob sich Moleküle in zylinderförmigen (Neuriten) oder kugelförmigen (Soma) Strukturen bewegen.
Zusammen ergeben diese Daten ein multiparametrisches Messverfahren, das ein wesentlich klareres Bild der zellulären Struktur liefert.
Was wir herausgefunden haben
Wir haben gesunde Probanden auf leistungsstarken 3T-MRT-Scannern untersucht und konnten:
- Beobachten, dass die Metabolitendiffusion mit längerer Messzeit langsamer und unregelmäßiger wurde – ein Zeichen dafür, dass Moleküle an Zellmembranen stoßen.
- Realistische Zellgrößen berechnen:
- Neuronale Somata ≈ 6–8 µm
- Gliale Somata ≈ 9 µm
- Neuriten-Durchmesser ≈ 1 µm
- Zeigen, dass die Kombination beider Methoden zu stabilen und physikalisch plausiblen Modellen führte – im Gegensatz zu früheren, teils widersprüchlichen Ergebnissen.
- Gleichzeitig Wasserdiffusion messen, die Austauschzeiten zwischen Kompartimenten von 13–17 ms ergab.
Warum das wichtig ist
Diese Methode verwandelt die MRT in ein „virtuelles Mikroskop“.
Sie könnte helfen, Krankheiten zu erkennen oder zu verfolgen, die die Zellstruktur verändern – etwa Alzheimer oder Multiple Sklerose – lange bevor Gewebeschäden sichtbar werden.
Und obwohl wir derzeit spezielle Hochleistungs-Scanner nutzen, lässt sich das Verfahren so anpassen, dass es auf klinischen MRT-Geräten in weniger als einer Stunde durchgeführt werden kann.
Blick nach vorn
Wir sind begeistert von dem, was das bedeutet: Eine MRT, die nicht nur zeigt, wo im Gehirn etwas passiert, sondern auch wie die Zellen selbst aufgebaut sind.
Das ist ein Schritt hin zu einer nichtinvasiven, zellspezifischen Bildgebung – und vielleicht eines Tages zu einer Methode, um neurologische Erkrankungen auf zellulärer Ebene zu diagnostizieren und zu überwachen.